TCGA数据库分析宫颈鳞状细胞癌(CESC)
TCGA数据库数据下载与整理,差异分析,lncRNA与mRNA分离,生存分析,ceRNA调控网络,miNRA差异分析,ID转换 [精华] 转自:http://www.dxy.cn/bbs/thread/36649535#36649535 |
|---|
生物信息学是将分子生物学与信息处理技术结合,以计算机为工具对生物信息进行储存、检索和分析的交叉学科,其目的是利用各种数据库,分析整理其数据的意义而揭示大量复杂的生物数据所赋予的生物学奥秘。癌症基因组图谱(TCGA)数据库是由美国国家癌症研究所(NCI)及国家人类基因组研究所(NHGRI)联合建立,其中包括丰富的数据类型和肿瘤类型,不需要任何费用即可获得大量数据,其次在TCGA下载的数据已经经过了前期繁琐的标准化处理,节省了人工处理时间和资源。 在分析之前,先把我们做出来的结果剧透给大家
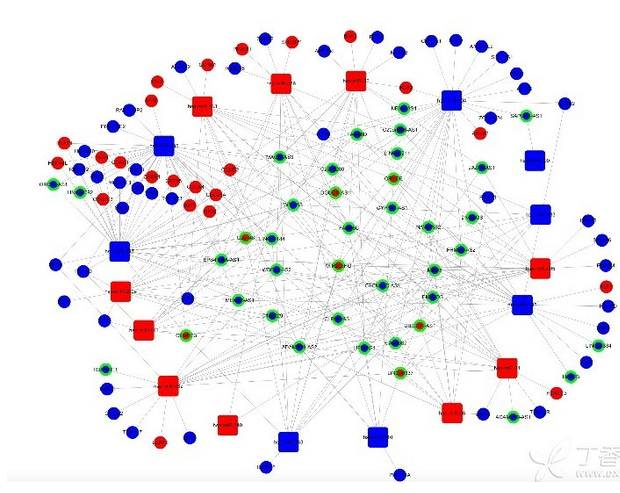
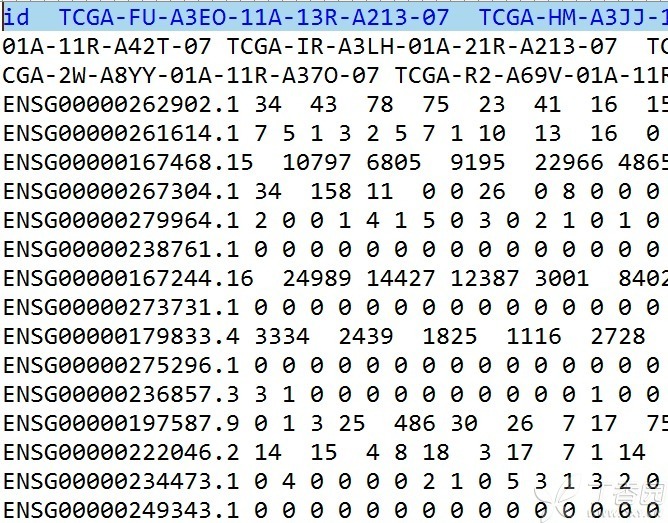
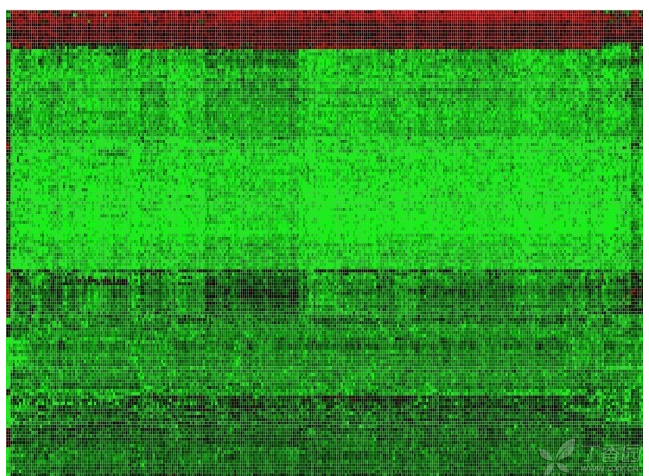
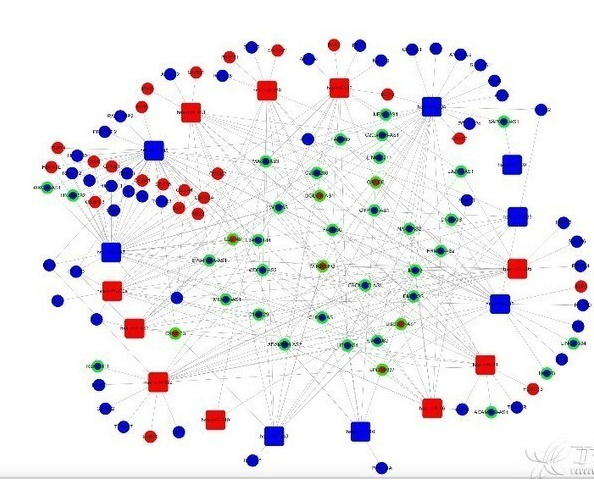
圆形代表DERAN,方块代表DEmiRNA,绿色外边圆形代表DElncRNA 分析GEO的朋友可以参考一下 分析GEO,并且在TCGA结合分析癌症的可以参考: GEO多芯片联合分析&TCGA数据库分析、生成分析对差异基因验证,论文&报告思路交流 一、 数据下载 使用TCGA数据库获取肿瘤和正常配对组织的基因数据及miRNA数据。得到3个正常样本和304个CESC肿瘤样本。分别做临床分析、mRNA差异分析、lncRNA差异分析、miRNA差异分析、生存分析、ceRNA调控网络构建。 二、 mRNA差异表达 1、如何得到矩阵文件? 下载的是counts文件,每个样本的压缩包保存在单独的文件中。 首先需要把所有的压缩包放在同一个文件夹内,统一解压,然后从几百个counts文件提取矩阵。得到Ensembl的矩阵,用Homo_sapiens.GRCh38.87.chr.gtf文件进行转换,得到symbol的矩阵,包括mRNA,和lncRNA及其他一起。 分别提取mRNA的矩阵和lncRNA的矩阵。 2、使用R的edgeR包,筛选条件|logFC|>2 & FDR<0.01 得到1933个差异基因,1195个下调,738个上调,部分差异基因如下表 3、用heatmap包得到前100上调差异基因和前100下调差异基因的热图
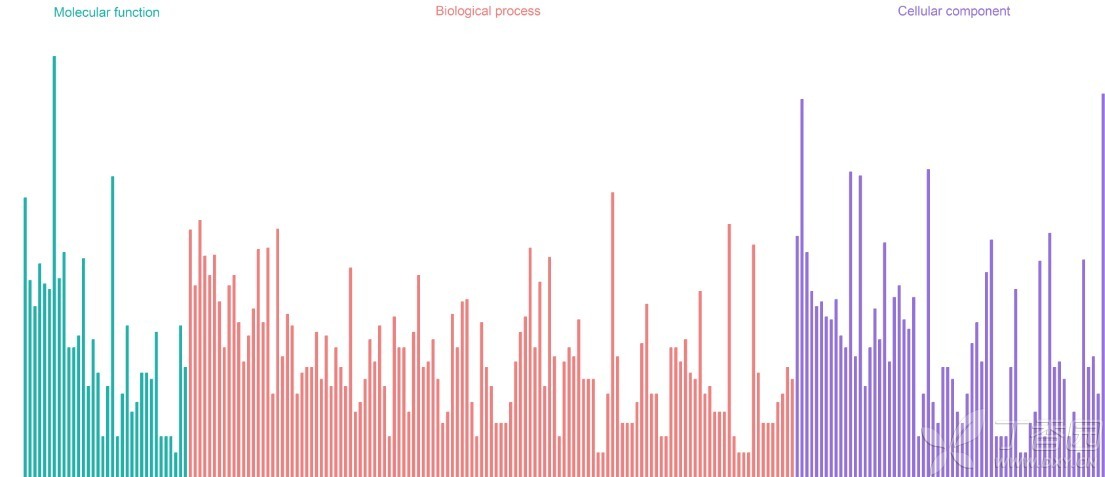
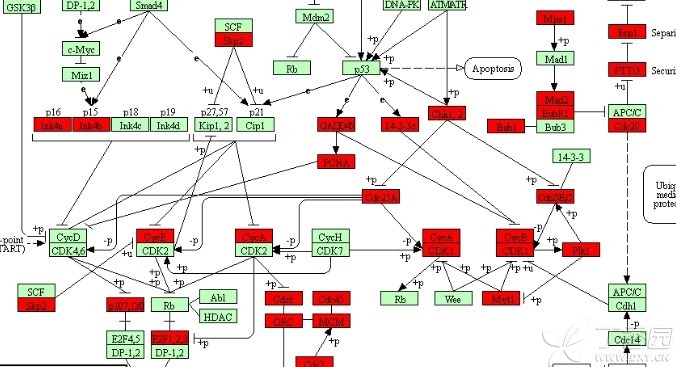
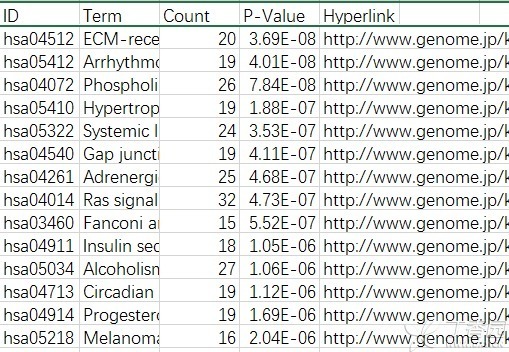
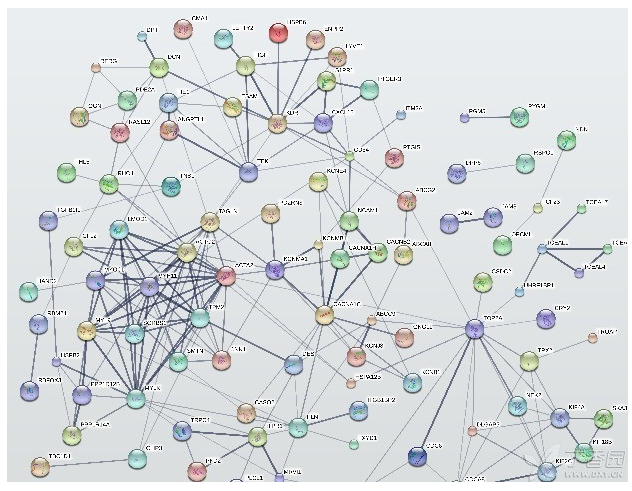
4、GO功能分析 DAVID在线工具分析所有差异基因的GO功能,筛选条件PValue<0.01,得到223个GO。用R做柱状图得到GO功能分析图: 5、KEGG分析 对差异差异基因做KEGG分析,使用的是KOBAS 3.0,这是一款简单容易操作的在线分析工具,需要注意的是,KOBAS在线工具需要输入的是Entrez Gene ID,而我们得到的差异基因是Gene ID,这个需要转换,转换的工具很多,我们选择DAVID在线工具做转换。结果可以得到KEGG通路图和详细的表,筛选条件P-Value<0.01,得到67个KEGG通路,其中一条通路如下图: 6、蛋白互作网络 蛋白互作网络在论文出现的次数没有以前频繁,不过要看文章研究的重点和方向。我们选择String软件作为研究工具,这款可视在线工具使用非常简单,需要注意的是输入的Gene ID不能超过2000,输出PNG时需要对图片进行调整,调整有很多参数可以选择,比如相关性、是否出现游离基因,如果图片很大很混乱,需要把相关性调大,一般情况下剔除游离基因。 得到下面的蛋白互作网络:
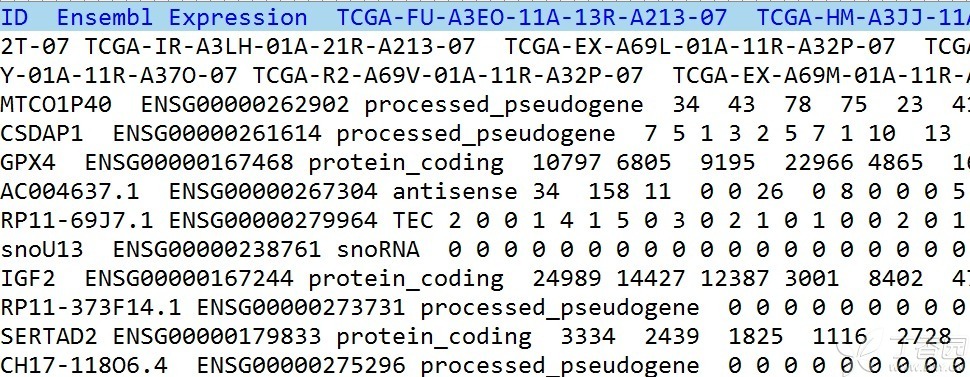
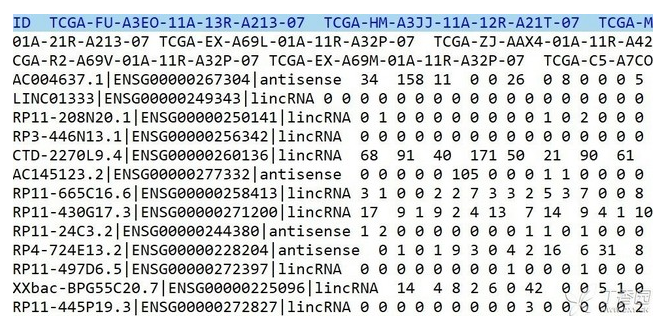
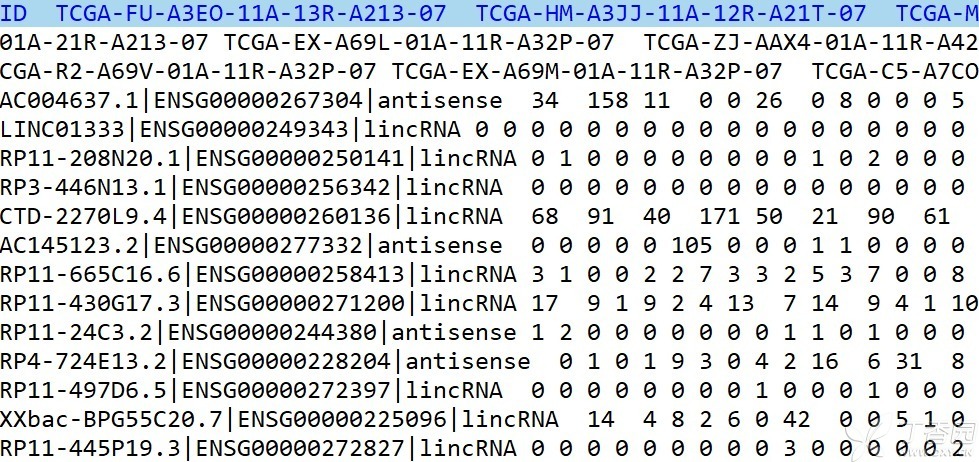
三、lncRNA差异表达 1、获得lncRNA矩阵 TCGA数据库下载的转录数据,包含mRNA和lncRNA,是在同一部分文件中,提取lncRNA矩阵选取antisense、lincRNA、sense_intronic等。
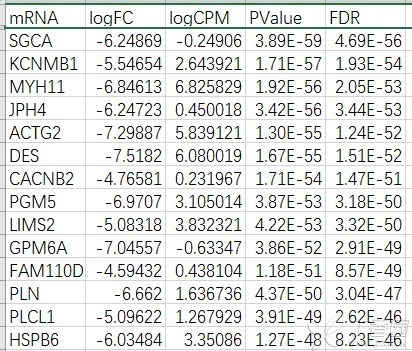
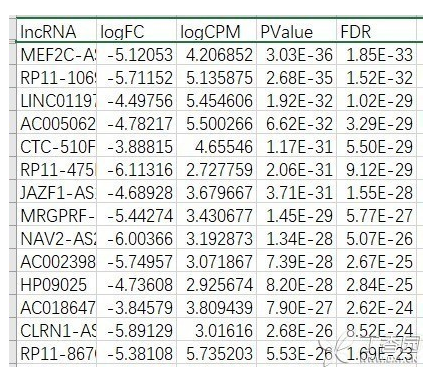
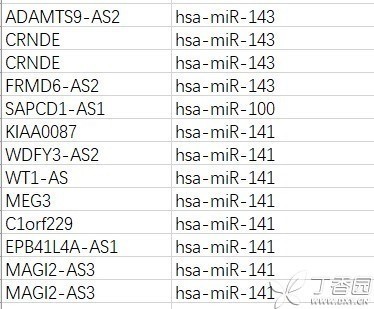
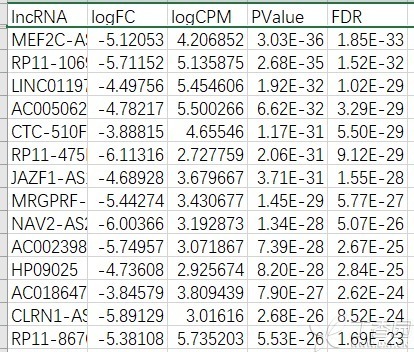
2、使用edgrR包,筛选条件|logFC|>2 & FDR<0.01,得到494个差异lncRNA,其中下调360个,上调134个,部分差异lncRNA如下表
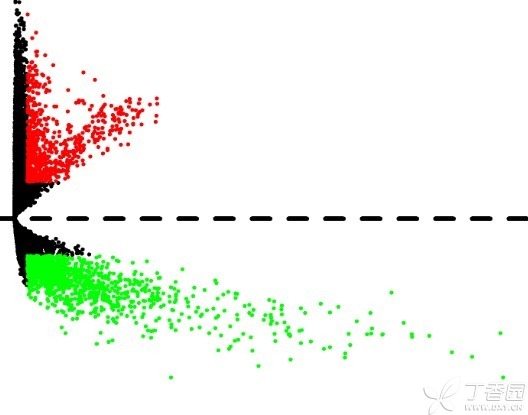
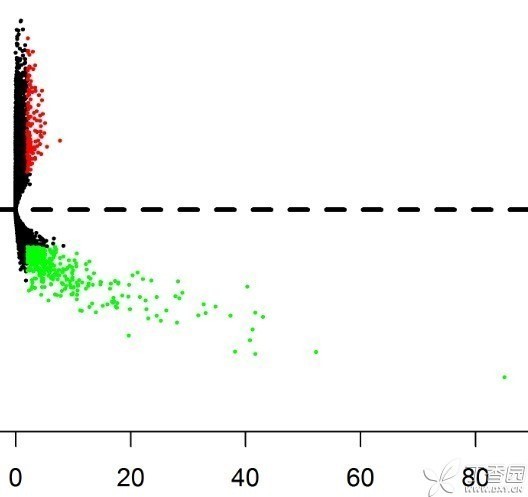
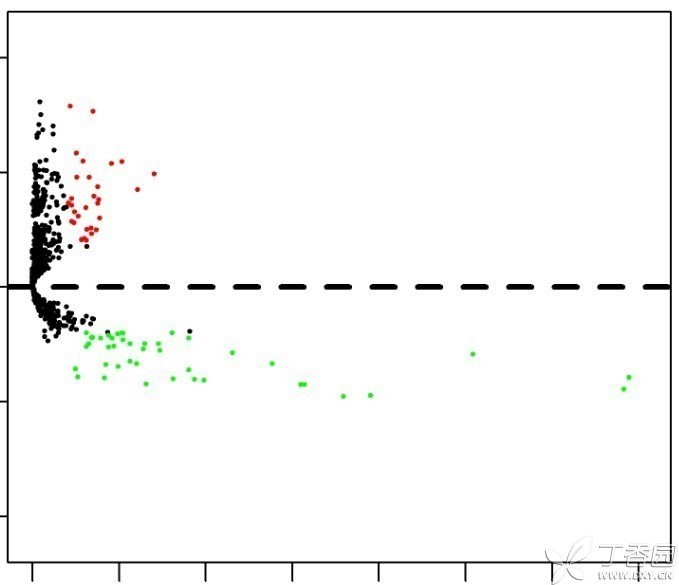
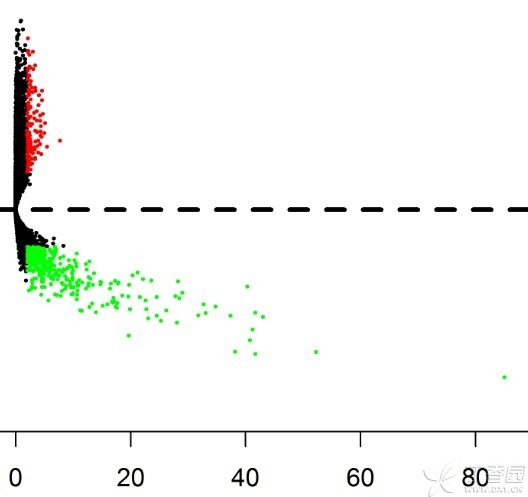
火山图 3、上调前100个,下调前100个lncRNA聚类做热图
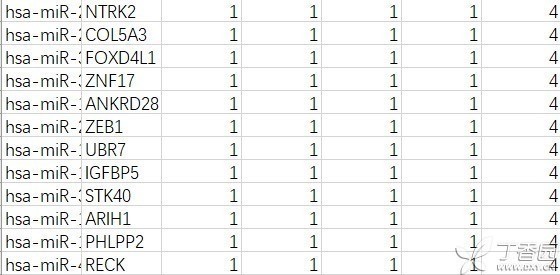
四、miRNA差异表达 1、首先需要获得miRNA的矩阵文件,从TCGA下载下来的是每个样本单独的矩阵文件,需要利用perl或者python脚本提取,提取得到需要进行分析的文本文件。 2、使用edgrR包,筛选条件|logFC|>2 & FDR<0.01,得到74个差异miRNA,其中下调43个,上调31个,部分差异miRNA如下表:
3、热图分析,分析和前面mRNA差不多。
五、ceRNA网络构建 1、用在线工具,比如StarBase,做lncRNA和miRNA的比对,这一步是构建ceRNA的关键。这里494个差异lncRNA和74个差异miRNA进行比对。得到39个DElncRNA和18DEmiRNA相互作用。 2、miRNA靶基因预测 利用TargetScan、miRDB、miRanda、miRTarBase在线工具对18个miRNA进行靶基因预测,得到的靶基因在去和差异基因做淘汰,得到69个靶基因也就是DEmRNA。 3、构建ceRNA网络 经过一次比对,一次预测,最终得到39个DElncRNA、18个DEmiRNA和69个DEmRNA,以及它们之间的相互关系。使用cytoscape对具有相关性的lncRNA、miRNA、miRNA靶基因进行可视化。就可以得到ceRNA网络。cytoscape的使用有很多学问,如何做出漂亮的图需要很多时间和审美。
希望这个帖子对大家有帮助 |
前辈,又来求教啦~
提取lncRNA矩阵选取antisense、lncRNA、sense_intronic等,这步是怎么提取的呢?我之前是手动在Excel表格中把LNC开头的找出,这样看来很不全面,想请前辈分享下具体的方法
后面会把所有分析过程分享给大家,ceRNA网络里面的lncRNA和miRNA需要做比对,miRNA的靶基因需要与差异基因做比对,这样才能得到DElncRNA,和DEmiRNA |






评论专区